Interamerican Journal of Heath Sciences 4 (2024) - ISSN 2953-3724
In silico exploration of potential breast cancer drugs
Exploración in silico de fármacos potenciales contra el cáncer de mama
Daneilys de Dios Hernández1 *
1Universidad de Ciencias Médicas Pinar del Río.
![]()
Received: 28-06-2023 Revised: 05-09-2023 Accepted: 03-01-2024 Published: 04-01-2024
How to Cite: de Dios Hernández D. In silico exploration of potential breast cancer drugs. Interamerican Journal of Health Sciences. 2024; 4:171. https://doi.org/10.59471/ijhsc2024171
ABSTRACT
Introduction: breast carcinoma is the most common neoplasm among women. One of the current problems is the emergence of drug resistance. Cathepsin B is a cysteine protease that is overexpressed in tumor tissue. The search for new therapeutic alternatives derived from the nitroxoline nucleus constitutes an encouraging solution against the disease.
Objective: to evaluate in silico potential cathepsin B inhibitors as therapeutic targets in the treatment of breast carcinoma.
Methods: from the PubChem database, 12 ligands derived from the nitrosoline nucleus were obtained, which were converted to 3D and their energy was minimized by applying the GAFF force field. The specific characteristics of the protease active site were determined with the Proteins Plus web server to carry out molecular docking studies with Autodock Tools.
Results: ligands 1511784, 14125599 and 45487202 showed a favorable affinity energy (ΔG=-5,56; -5,51 and -5,08 respectively) and inhibition constant by inhibiting key residues of the catalytic site of cathepsin B. The amino acids were: His199, His110, His111, Gln23 and Gly198. The main interaction was by hydrogen bonding.
Conclusions: nitrosoline contains anticancer properties. Ligands 1511784, 14125599 and 45487202 constitute potential drugs against breast cancer. Therefore, in silico analyzes reduce the cost of current research and contribute to the specificity and immunogenicity of therapies and pharmacological biosafety.
KEYWORDS
Nitrosoline, Cathepsin B Inhibitors, Molecular Docking.
RESUMEN
Introducción: el carcinoma de mama es la neoplasia más frecuente entre las mujeres. Uno de los problemas actuales es la resistencias a fármacos. La catepsina B es una cisteíno proteasa que se sobre expresa en el tejido tumoral. La búsqueda de nuevas alternativas terapéuticas derivadas del núcleo de la nitrosolina constituye una solución contra la enfermedad.
Objetivo: evaluar in silico inhibidores potenciales de la catepsina B como blancos terapéuticos en el tratamiento del carcinoma mamario.
Métodos: a partir de la base de datos PubChem se obtuvieron 12 ligandos derivados del núcleo de la nitrosolina que fueron convertidos a 3D y se les minimizó la energía a partir de la aplicación del campo de fuerza GAFF. Se determinaron las características del sitio activo de la proteasa con el servidor web Proteins Plus para la realización de los estudios de acoplamiento molecular con Autodock Tools.
Resultados: los ligandos 1511784, 14125599 y 45487202 mostraron una energía de afinidad (ΔG=-5,56; -5,51 y -5,08 respectivamente) y constante de inhibición favorable al inhibir residuos claves del sitio catalítico de la catepsina B. Los aminoácidos fueron: His199, His110, His111, Gln23 y Gly198. La principal interacción fue por enlace de hidrógeno.
Conclusiones: la nitrosolina contiene propiedades anticancerígenas. Los ligandos 1511784, 14125599 y 45487202 constituyen fármacos potenciales contra el cáncer de mama. Por ello, los análisis in silico reducen el costo de las investigaciones y contribuyen a la especifidad e inmunogenicidad de las terapias y la bioseguridad farmacológica.
PALABRAS CLAVES
Nitrosolina, Inhibidores de la Catepsina B, Acoplamiento Molecular.
INTRODUCCIÓN
El carcinoma de mama (CM) es la neoplasia más frecuente entre las mujeres. Constituye la segunda causa de muerte por cáncer entre mujeres, después del cáncer de pulmón.(1) Se estima que el 30 % de las neoplasias mamarias se deben a factores de riesgo modificables, como exceso de peso corporal, inactividad física y la ingesta de alcohol.(2) Ocurre cuando las células de la glándula mamaria crecen de forma descontrolada debido al fallo de los mecanismos regulatorios que controlan su proliferación.(3) Las tasas de incidencia del cáncer de mama entre los años 2010 y 2019 aumentaron un 0,5 % anual.(2) Según el anuario estadístico, durante el 2019, se registraron en Cuba 4351 nuevos casos de CM. El grupo etario más afectado por el carcinoma de mama se ubicó entre 60-79 años, con 804 defunciones, lo que ratificó que, a mayor edad, mayor es el porciento de incidencia de esta afección.(4)
Las variantes genéticas asociadas a un mayor riesgo de CM implican mutaciones en los genes BRCA1/2. La terapia más ampliamente usada incluye los moduladores selectivos de los receptores de estrógeno tamoxifeno y raloxifeno, o los inhibidores de la aromatasa: anastrozol, letrozol y exemestano, para tratar a mujeres posmenopáusicas de 35 años o más.(1) Hasta el 80 % de los cánceres de mama invasivos son carcinomas ductales infiltrantes (CID).(5)
Uno de los problemas actuales de la oncología es la aparición de resistencias a un tratamiento, donde las células malignas se vuelven insensibles a los fármacos.(6) Actualmente el uso de herramientas computacionales aplicadas al diseño de fármacos (in silico), permite la búsqueda de nuevas terapias no exploradas en el diseño experimental. Ello hace posible la selectividad y eficacia de una terapia, lo que reduce el tiempo y costo de la investigación presente.
La catepsina B es una proteasa lisosomal de tipo cisteíno y desempeña un papel importante en la proteólisis intracelular. Su sobre expresión ha sido observada en cáncer de esófago, gástrico, próstata, glioblastoma y cáncer de seno. Por ello, es considerada como un blanco potencial con propiedades terapéuticas debido a su amplia participación en la progresión del cáncer.(7,8) Actualmente se trabaja en el diseño de inhibidores de la catepsina B y se ha observado una reducción en la tumurogénesis. El uso de inhibidores derivados de la nitrosolina constituye fármacos potenciales en el tratamiento de tumores.(9) A continuación se trabajan compuestos derivados del núcleo de la nitrosolina, así como estudios de acoplamiento molecular que permitan la selección de posibles candidatos para el tratamiento del carcinoma mamario.
MÉTODOS
Se realizó una búsqueda de compuestos derivados y similares a la nitroxolina en la base de datos PubChem (https://pubchem.ncbi.nlm.nih.gov/). Se encontraron 19 compuestos, 93 sustancias y 390 compuestos relacionados estructuralmente. Posteriormente se realizó un filtrado en PubChem, teniendo en cuenta características como: peso molecular, cantidad de átomos de carbono, número de rotaciones, y el año cuando fue anotado el compuesto. Para este último parámetro, se escogieron los últimos 5 años. Como resultado se obtuvieron un set de datos de 30 compuestos con las características deseadas.
El set de datos obtenido en 2D fue convertido a 3D (estructura tridimensional del compuesto) mediante el programa Avogadro, versión 1.2.0. A cada compuesto se le optimizó su geometría y se le aplicó el campo de fuerza GAFF (General AMBER Force Field). Este tipo de campo de fuerza es compatible con moléculas orgánicas que presenten en su estructura C, H, O, N, S, P, F, Cl y Br. De los 30 compuestos analizados, aquellos que presentaron un valor de estabilidad energética mayor a 100 KJ/mol fueron desechados. Como resultado, la data de sustancias se redujo a 12 compuestos. La visualización molecular de cada compuesto fue posible con el software PoV Ray 3.7.
La estructura 3D de la proteína receptora de interés fue tomada del Protein Data Bank (PDB, https://www.rcsb.org/). Se escogió la estructura 8b5f. Se usó el servidor web Proteins Plus (https://proteins.plus), una herramienta computacional para el estudio de las interacciones ligando-proteína. Ello permitió seleccionar el tamaño del bolsillo de unión de la catepsina B, los residuos de aminoácidos potenciales del sitio activo y sus posibles interacciones para los estudios de acoplamiento molecular.
Seguidamente, la proteína fue separada de los ligandos que poseía, así como las moléculas de agua, para su procesamiento mediante el programa Pymol 1.74. Posteriormente, se realizaron estudios de acoplamiento molecular para estimar la interacción energética entre la catepsina B y cada uno de los ligandos, a partir del programa Autodock Tools 1.5.7.
RESULTADOS
A continuación, se muestran los compuestos derivados del núcleo de la nitrosolina extraídos de la base de datos PubChem y convertidos a 3D con el software Avogadro (Figura 1). En cada compuesto se estimó la fuerza entre sus átomos, lo que se evidencia a partir de un valor de energía potencial que se le asigna a cada uno. La energía está dada en KJ/mol (Tabla 1). Adicionalmente, la tabla contiene la fórmula química y el peso molecular de cada sustancia, así como el número de átomos y enlaces presentes en cada uno.
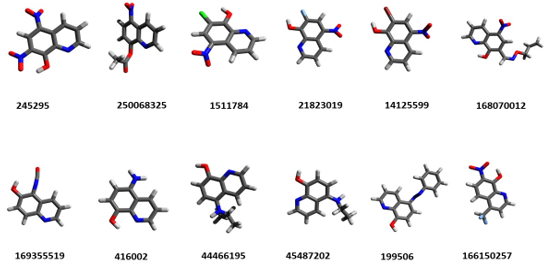
Figura 1. Compuestos derivados de la nitrosolina extraídos de la base de datos PubChem.
Se usó el programa PoV Ray para la visualización molecular 3D. Se muestran 12 compuestos en la representación de barras y esferas. Los átomos de carbono (C) se señalan de color gris; los hidrógenos (H), blanco; de rojo se señala el oxígeno (O); en azul oscuro, el nitrógeno (N); en verde, el cloro (Cl) y en azul claro, el flúor (F).
Tabla 1. Compuestos extraídos de PubChem
|
Ligando |
Nombre |
Fórmula molecular |
Energía (KJ/mol) (GAFF) |
Peso molecular (g/mol) |
No. átomos |
No. enlaces |
|
250068325 |
Nitrosolina |
C11H8N2O4 |
64,98 |
232,19 |
25 |
26 |
|
199506 |
5-fenil azo-8-hidroxiquinolina |
C15H11N3O |
62,75 |
249,267 |
30 |
32 |
|
245295 |
5,7-Dinitro-8-quinolinol |
C9H5N3O5 |
14,92 |
235,153 |
22 |
23 |
|
416002 |
5-Amino-8-hidroxiquinolina |
C9H8N2O |
45,78 |
160,173 |
20 |
21 |
|
1511784 |
7-cloro-5-nitroquinolina-8-ol |
C9H5ClN2O3 |
44,18 |
224,601 |
20 |
21 |
|
14125599 |
7-bromo-8-hidroxi-5-nitroquinolina |
C9H5BrN2O3 |
46,64 |
269,052 |
20 |
21 |
|
21823019 |
7-Flúor-5-nitroquinolina-8-ol |
C9H5FN2O3 |
34,02 |
208,146 |
20 |
21 |
|
44466195 |
5 Isopropilaminoquinolin-8-ol |
C12H14N2O |
78,48 |
202,252 |
29 |
30 |
|
45487202 |
5 Etilaminoquinolin-8-ol |
C11H12N2O |
81,42 |
188,226 |
26 |
27 |
|
166150257 |
4-Difluorometil-7-nitroquinolin-8-ol |
C10H6F2N2O3 |
95,08 |
240,163 |
23 |
24 |
|
168070012 |
5-nitro-7-prop-2-enoxi-imino-metilquinolina-8-ol |
C13H11N3O4 |
14,41 |
273,244 |
31 |
32 |
|
169355519 |
5-Isocianatoquinolin-6-ol |
C10H6N2O2 |
30,19 |
186,167 |
20 |
21 |
|
Se incluye los valores de energía calculado con el programa Avogadro 1.2.0. Se aplicó el campo de fuerza GAFF |
||||||
El uso del servidor web Proteins Plus permitió la caracterización estructural del bolsillo de unión de la catepsina B implicado en la catálisis. Predice los aminácidos claves del sitio catalítico y muestra los involucrados en la actividad catalítica de la enzima. Los resultados fueron tomados en cuenta para la realización de los estudios de acoplamiento molecular. Mediante el programa Autodock Tools fueron evaluados 12 ligandos como posibles inhibidores de la catepsina B. En la Tabla 2 se relacionan los resultados obtenidos.
Tabla 2. Resultados del acoplamiento molecular con el uso de Autodock Tools.
|
Ligando |
ΔG (KJ/mol) |
Ki (µM) |
Aminoácidos claves inhibidos |
E. electrostática (KJ/mol) |
E. torsional |
|
250068325 |
-5,64 |
- |
- |
0,33 |
0,89 |
|
199506 |
-7,27 |
4,69 |
His110, His111, Gly198 |
0,09 |
0,89 |
|
245295 |
-3,82 |
1,59 |
Gly198 |
1,05 |
0,89 |
|
416002 |
-4,79 |
307,43 |
His111, His199 |
0,04 |
0,6 |
|
1511784 |
-5,56 |
83,56 |
His111, His199, Gly198 |
0,24 |
0,6 |
|
14125599 |
-5,51 |
92,05 |
His111, His199, Gln23 |
0,37 |
0,6 |
|
21823019 |
-5,24 |
- |
- |
0,29 |
0,6 |
|
44466195 |
-5,39 |
- |
- |
0,15 |
0,89 |
|
45487202 |
-5,08 |
187,08 |
His111, His199, Gly198 |
0,04 |
0,89 |
|
166150257 |
-5,99 |
48,08 |
His199, Gln23, Gly198 |
0,27 |
0,89 |
|
168070012 |
-4,33 |
674,1 |
His111, His199 |
0,52 |
0,6 |
|
169355519 |
-5,26 |
140,27 |
His111, Gln23 |
0,09 |
0,6 |
|
Se muestra el valor de la energía de afinidad ligando-receptor. Solo se tuvo en cuenta los aminoácidos del sitio activo que eran inhibidos por cada compuesto. Se resalta en color rojo los resultados que se consideran potenciales fármacos que inhiben la catepsina B. ΔG representa la afinidad ligando-proteína y Ki, la constante de inhibición. |
|||||
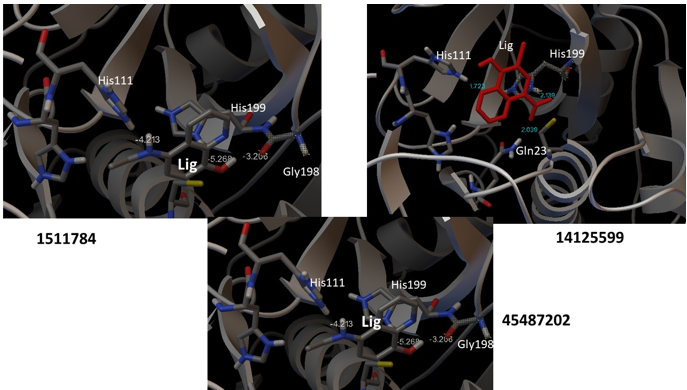
Nota: Se muestran los valores de energía en color blanco y en color verde, las distancias de enlace entre los átomos involucrados en la interacciones. Lig: representa el ligando; His: histidina; Gly, glicina y Gln, glutamina.
Figura 2. Interacción ligando-receptor para los compuestos 1511784, 14125599 y 45487202.
De la data de 12 compuestos, se seleccionaron los posibles inhibidores de la catepsina B teniendo en cuenta si inhibían aminoácidos claves y presentaran una constante de inhibición razonable. Los compuestos que cumplían los criterios anteriores fueron: 1511784, 14125599 y 45487202. Se muestran las interacciones por puentes de hidrógeno que se establecen entre estos ligandos y los aminoácidos del sitio activo de la catepsina B (Figura 2), así como las distancias entre estos enlaces. Adicionalmente se señala el valor de energía asociado a cada interacción.
A partir del uso de la herramientas de Autodock Tools se construyeron las principales interacciones entre los aminoácidos del bolsillo de unión de la catepsina B y los ligandos de interés (Figura 3). En cada caso el ligando se visualizan como una esfera sólida transparentes ubicado en el centro y los aminoácidos en forma de tubos. Las interacciones que se establecen se muestran como esferas de celdas.
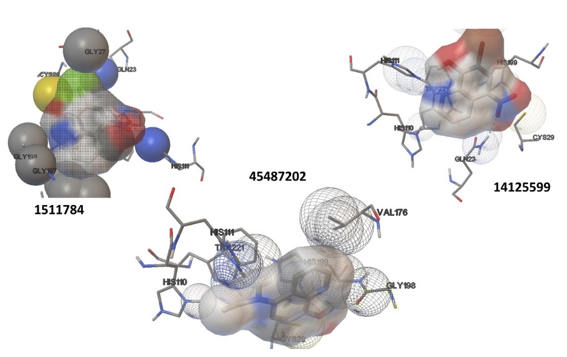
Nota: Las interacciones se muestran como esferas de celdas. En el centro se muestra cada ligando.
Figura 3. Interacciones presentes entre los ligandos 1511784, 14125599 y 45487202 y los aminoácidos claves para el proceso de catálisis
DISCUSIÓN
La catepsina B es una proteasa cisteíno lisosomal involucrada en la progresión tumoral y metástasis. Representa un objetivo crucial para el desarrollo de nuevos agentes antitumorales. Los inhibidores de proteasas que se sobreexpresan en diferentes carcinomas están tomando auge actualmente. Especialmente, el uso de compuestos no peptídicos con actividad inhibitoria en modelos in silico e in vitro, ha resultado de utilidad contra la progresión tumoral. (9) Se ha descubierto que la nitroxolina tiene propiedades anticancerígenas eficaces en cáncer de mama, glioma, cáncer de vejiga, páncreas y de próstata. (10,11) Está implicada en la activación de la apoptosis celular, la detención del ciclo celular y la supresión de la angiogénesis. Estudios en modelos animales han demostrado que la nitroxolina es un inhibidor eficaz y reversible de la actividad catalítica de la catepsina B. (12,13)
En la Figura 1 se observa la data de compuestos extraídos de la base de datos de PubChem. Todos son derivados del núcleo de la nitrosolina y se les minimizó la energía, de modo que se encontraran en una conformación estable, análoga a su forma natural. Se le aplicó el campo de fuerza GAFF, compatible para posibles fármacos que tengan en su estructura C, H, O, N, F, Cl, entre otros elementos. En este sentido, en la Tabla 1 se relacionan los valores de energía de cada compuesto que fue sometido al campo GAFF y su fórmula molecular correspondiente. Todos los compuestos seleccionados tuvieron entre 9 y 15 carbonos.
Cada ligando se evaluó en 10 conformaciones distintas en el sitio de la catpesina B. Sin embargo, se tomó el mejor valor en cada caso. La afinidad ligando-proteína está dada por una función de puntuación que implica la suma de las energías electrostática y de Van der Waals. A menor valor de afinidad, más estable será el compuesto. Todos los valores se ubicaron por debajo de “0”, lo que indica que el complejo ligando- receptor sea estable y con mayor posibilidad de ocurrencia de la interacción. Sin embargo, los compuestos 250068325, 21823019 y 44466195 fueron desestimados (Tabla 2), pues no inhiben ningún residuo aminoacídico del sitio catalítico de la catepsina B. Los ligandos 245295, 416002 y 168070012 presentaron valores de energía menores con respecto al resto, lo que indica una menor estabilidad de la interacción con el sitio activo del receptor.
En este sentido, los fármacos potenciales seleccionados fueron 1511784, 14125599 y 45487202, pues ambos presentan afinidades de acoplamiento (ΔG=-5,56; -5,51 y -5,08 respectivamente) por la catepsina B e inhiben aminoácidos claves de su bolsillo de unión: Gln23, His 111, His199 y Gly 198. Los resultados son similares a otros estudios in silico reportados para la catepsina B en cancer y la enfermedad de Alzheimer. En este la energía de afinidad para los inhibidores fue -6.74 y -6.12 (KJ/mol).(14,15) La His 199 es un aminoácido esencial en la catálisis de la tríada catalítica de las cisteíno proteasas, que están formadas por Asn, Cys e His. La Cys y la His forman un par iónico en la díada catalítica. La His actúa junto a la cisteína en la formación del par nucleofílico tiolato (grupo tiol de la cisteína)-imidazol (grupo de la His). La Gln23 participa posteriormente, en la estabilización del intermediario tetraédrico a partir de la formación de un puente de hidrógeno.(16)
En la tabla 2 se muestra la constante de inhibición (Ki) para los ligandos que inhibieron algún aminácido del sitio catalítico. Los valores fueron de 83,56; 92,05 y 187,08 µM para los inhibidores potenciales 1511784, 14125599 y 45487202. Existen diferencias entre la Ki calculada y experimental, sin embargo, este valor in silico nos da una medida de la inhibición competitiva, no reversible que podrían tener estos compuesto en ensayos in vitro.
En la Figura 2 se muestran las interacciones por puentes de Hidrógeno que se establecen entre cada ligando y la catepsina B, así como la energía de afinidad de cada interacción. En todos los casos el valor de ΔG es negativo lo que evidencia la estabilidad de unión del complejo formado. En el caso del ligando 14125599 se reporta la distancia de enlace H-O, donde los valores están entre 1,6 a 2,0 Å. Ello indica que el ligando se comporta como aceptor de hidrógenos, mientras que el aminoácido del sitio activo, corresponde al donor de O o N.
En la figura 3 se muestran las interacciones que se establecen entre ligando-receptor. La His 111 es responsable de la actividad dipeptidil carboxipeptidasa y actúa junto a la His 110 en la estabilización del extremo carboxílico del péptido sustrato. La His se ubica de modo adyacente a residuos pequeños como Gly o alanina (Ala). En este caso, se observa en la Figura 2, ligando 1511784, la posición adyacente de la His119 respecto a la Gly198. Los candidatos farmacológicos 1511784, 14125599 y 45487202 forman enlaces de hidrógeno con estos resisuos de aminoácido, lo que sugiere su papel inhibidor en la catepsina B contra el cáncer de mama. La técnica de acoplamiento presenta algunas limitaciones pues considera al receptor (catepsina B) como una estructura rígida durante la interacción. Presenta una baja sensibilidad para moléculas grandes y un máximo de 8 enlaces rotables.(17) En este caso se analizaron moléculas pequeñas que tenían menos de 5 enlaces rotables.
Los resultados de este estudio computacional sugieren que los inhibidores 1511784, 14125599 y 45487202 presentan afinidades de acoplamiento por el sitio activo de la catepsina B con valores de energía libre de interacción favorables. Se evidencia la inhibición de residuos claves para la función de la catepsina B. Ello contribuye a la búsqueda de nuevas estrategias para combatir el cáncer de mama, específicamente en la detección de moléculas derivadas del núcleo de la nitrosolina. Las simulaciones realizadas, pesar de presentar resultados altamente predictivos, desestiman el modo y tiempo de acción de las moléculas, así como las concentraciones óptimas de actividad lo cual depende exclusivamente de ensayos experimentales.
REFERENCIAS BIBLIOGRÁFICAS
1. Trayes KP, Cokenakes SE. Breast Cancer Treatment. American Family Physician. 2021 Aug; 104(2):171-8. Disponible en: https://www.aafp.org/pubs/afp/issues/2021/0800/p171.pdf
2. Giaquinto AN, Sung H, Miller KD, Kramer JL, Newman LA, Minihan A, et al. Breast Cancer Statistics. CA Cancer J Clin. 2022 Dec; 72(6):524–18. Disponible en: https://acsjournals.onlinelibrary.wiley.com/doi/pdf/10.3322/caac.2175
3. Waks A, Winer E. Breast cancer treatment a review. Rev Clinical Review and Education. 2019; 321(3): 3p. Disponible en: http://bdrc.tums.ac.ir/uploads/140/2020/Jun/17/Breast-Cancer-Treatment-Jan-2019-1.pdf
4. Ministerio de Salud Pública. Anuario estadístico 2019 La Habana. MINSAP. Disponible en: https://instituciones.sld.cu/ucmvc/files/2023/10/Anuario-Estad %C3 %ADstico-de-Salud-2022-Ed-2023.pdf
5. Watkins EJ. Overview of breast cancer. Journal of the American Academy of Pas. 2019 Oct; 32(10):13-5. Disponible en: https://journals.lww.com/jaapa/fulltext/2019/10000/Overview_of_breast_cancer.3.aspx
6. De Dios DH. Selección de blanco terapéutico en la catepsina B para el desarrollo de fármacos contra el cáncer de mama. Rev Ciencias Médicas. 2021; 25(5): 10p. Disponible en: http://revcmpinar.sld.cu/index.php/publicaciones/article/view/5003
7. Aggarwal N, Sloane B. Cathepsin B: Multiple roles in cancer. Rev Proteomics Clin. Appl. 2014; 8(5): 427-11. Disponible en: https://onlinelibrary.wiley.com/doi/abs/10.1002/prca.201300105 7.
8. Gondi C, Rao J. Cathepsin B as a cancer target. Rev Expert Opin Ther Targets. 2013; 17(3): 281–11. Disponible en: https://www.ncbi.nlm.nih.gov/pmc/articles/PMC3587140/
9. Sosic I, Mitrovic´ A, Hrvoje C, Knez D, Brodnik HZ, Štefane B et al. Cathepsin B inhibitors: Further exploration of the nitroxoline core. Bioorganic & Medicinal Chemistry Letters. 2018; 28: 1239–9. Disponible en: https://www.sciencedirect.com/science/article/abs/pii/S0960894X18301471
10. Xu N, Huang L, Li X, Watanabe M, Li C, Xu A et al. The Novel Combination of Nitroxoline and PD-1 Blockade, Exerts a Potent Antitumor Effect in a Mouse Model of Prostate Cancer. International Journal of Biological Sciences. 2019; 15(5): 919-9. Disponible en: https://www.ncbi.nlm.nih.gov/pmc/articles/PMC6535792/
11. Xu N, Lin W, Sun J, Sadahira T, Xu A, Watanabe M et al. Nitroxoline inhibits bladder cancer progression by reversing EMT process and enhancing anti-tumor immunity. Journal of Cancer. 2020; 11(22): 6633-6641.
12. Mirkovic B, Markelc B, Butinar M, Mitrovic A, Sosic I, Gobec S, et al. Nitroxoline impairs tumor progression in vitro and in vivo by regulating cathepsin B activity. Oncotarget. 2015; 6: 19027-42. Disponible en: https://www.ncbi.nlm.nih.gov/pmc/articles/PMC4662473/
13. Mitrovic A, Kljun J, Sosic I, Ursic M, Meden A, Gobec S et al. Organoruthenated Nitroxoline Derivatives Impair Tumor Cell Invasion through Inhibition of Cathepsin B Activity. Inorganic Chemistry. 2019; 58(18): 12334-12347. Disponible en: https://pubs.acs.org/doi/full/10.1021/acs.inorgchem.9b01882
14. Ranjbar D, Reza M, Dehghani Z, Firuzi D, Edraki N, Khoshneviszadeh M. Dihydronaphthalenone chalconoid derivatives as potential cathepsin B inhibitors; design, synthesis, cytotoxicity evaluation and docking analysis. Braz. J. Pharm. Sci. 2021;57: 1-14. Disponible en: https://www.scielo.br/j/bjps/a/GTb8RkyrDwSRSbKCpRBnSMK/?lang=en
15. Chitranshi N, Kumar A, Sheriff S, Gupta V, Godinez A, Saks D et al. Identification of Novel Cathepsin B Inhibitors with Implications in Alzheimer’s Disease: Computational Refining and Biochemical Evaluation. Cells. 2021; 10(1946). Disponible en: https://www.mdpi.com/2073-4409/10/8/1946
16. Naranjo F. Análisis in silico de la catepsina B de Fasciola hepática como diana terapéutica. REDVET [Internet]. 2009 [citado: 08/03/2021]; 10(2): 1-36. Disponible en: https://www.redalyc.org/pdf/636/63617114011.pdf
17. Guadalupe W, Paucara B, Grados, R. Acomplamiento molecular: criterios prácticos para la selección de ligandos biológicamente activos e identificación de nuevos blancos terapéuticos. Rev.Cs.Farm. y Bioq. 2019; 7(2). Disponible en: http://www.scielo.org.bo/scielo.php?pid=S2310-02652019000200006&script=sci_arttext
FINANCIACIÓN
La autora no recibió financiación para el desarrollo de la presente.
CONFLICTO DE INTERESES
La autora declara que no existe conflicto de intereses.
CONTRIBUCIÓN DE AUTORÍA
Conceptualización: Daneilys de Dios Hernández.
Redacción –borrador inicial: Daneilys de Dios Hernández.
Redacción –revisión y edición: Daneilys de Dios Hernández.